Biosensore
Un biosensore è un dispositivo analitico, utilizzato per il rilevamento di una sostanza chimica, che combina un componente biologico con un detector fisico-chimico .[1][2][3][4] L'elemento biologico sensibile all'analita, ad esempio tessuto, microrganismi, organelli, recettori cellulari, enzimi, anticorpi, acidi nucleici, ecc., è un materiale di origine biologica o un componente biomimetico che interagisce, si lega o riconosce l'analita in studio. Gli elementi biologicamente sensibili possono anche essere prodotti con l'ingegneria biologica . Il trasduttore o l'elemento rivelatore, che trasforma un segnale in un altro, genera un segnale fisico-chimico: ottico, piezoelettrico, elettrochimico, elettrochemiluminescenza ecc., risultante dall'interazione dell'analita con l'elemento biologico, per misurarlo e quantificarlo facilmente. Il dispositivo di lettura del biosensore si collega al sistema elettronico associato o ai processori di segnale che sono in primo luogo responsabili della visualizzazione intuitiva dei risultati .[5] Questo a volte rappresenta la parte più costosa di tutto il dispositivo sensore, tuttavia è possibile generare un display intuitivo che includa sia il trasduttore che l'elemento sensibile ( sensore olografico ). I lettori progettati e prodotti su misura per adattarsi ai diversi principi di funzionamento dei biosensori.
Biosensore come sistema
[modifica | modifica wikitesto]Un biosensore è tipicamente costituito da un biorecettore (enzima/anticorpo/cellula/acido nucleico/aptamero), un componente trasduttore (materiale semiconduttore/nanomateriale) e un sistema elettronico che include un amplificatore di segnale, un processore e un display.[6] I trasduttori e l'elettronica possono essere combinati, ad esempio, in sistemi di microsensori basati su CMOS .[7][8]. L'elemento di riconoscimento, spesso chiamato biorecettore, utilizza biomolecole di organismi o recettori modellati su sistemi biologici per interagire con l'analita di interesse. Questa interazione è misurata dal biotrasduttore che emette un segnale misurabile proporzionale alla presenza dell'analita target nel campione. L'obiettivo generale del design di un biosensore è consentire test rapidi e convenienti nel luogo di assistenza sanitaria in cui è stato acquisito il campione (Point of Care Testing).[1][9][10]
Biorecettori
[modifica | modifica wikitesto]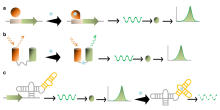
In un biosensore, il biorecettore è progettato per interagire con l'analita specifico di interesse per produrre un effetto misurabile dal trasduttore. Un'elevata selettività per l'analita rispetto a una matrice composta da altri componenti chimici o biologici è un requisito chiave del biorecettore. Sebbene il tipo di biomolecola utilizzata possa variare ampiamente, i biosensori possono essere classificati in base a tipi comuni di interazioni dei biorecettori che coinvolgono: anticorpi/antigene,[11] enzimi/ligandi, acidi nucleici/DNA, strutture cellulari/cellule o materiali biomimetici.[12][13]
Interazioni anticorpo/antigene
[modifica | modifica wikitesto]Un immunosensore utilizza l'affinità di legame molto specifica degli anticorpi per un composto o un antigene specifico. La natura specifica dell'interazione anticorpo-antigene è analoga a un adattamento di serratura e chiave; in quanto l'antigene si legherà all'anticorpo solo se ha la conformazione corretta. Gli eventi di legame provocano un cambiamento fisico-chimico che, in combinazione con un tracciante, come molecole fluorescenti, enzimi o radioisotopi, può generare un segnale. Esistono limitazioni nell'utilizzo di anticorpi nei sensori: 1. La capacità di legame dell'anticorpo dipende fortemente dalle condizioni del test (ad es. pH e temperatura); 2. l'interazione anticorpo-antigene è generalmente robusta, tuttavia, il legame può essere rotto da reagenti caotropici, solventi organici o persino radiazioni ultrasoniche.[14][15]
Le interazioni anticorpo-antigene possono essere utilizzate anche per test sierologici o per il rilevamento di anticorpi circolanti come reazione ad una specifica malattia. È importante sottolineare che i test sierologici sono diventati una parte importante nella risposta globale alla pandemia di COVID-19 .[16]
Proteine leganti artificiali
[modifica | modifica wikitesto]L'uso degli anticorpi come componente di bioriconoscimento dei biosensori presenta diversi inconvenienti. Gli anticorpi hanno pesi molecolari elevati e stabilità limitata, contengono legami disolfuro essenziali e sono costosi da produrre. Diversi approcci sono stati testati per superare queste limitazioni. In un approccio, frammenti di legame ricombinante (Fab, Fv o scFv) o domini (VH, VHH ) di anticorpi sono stati ingegnerizzati.[17] In un altro approccio, piccoli scaffold proteici sono stati progettati con proprietà biofisiche favorevoli per generare famiglie artificiali di proteine leganti l'antigene (AgBP), in grado di legarsi in modo specifico a diverse proteine target pur mantenendo le proprietà favorevoli della molecola madre. Gli elementi della famiglia che si legano in modo specifico a un determinato antigene bersaglio, sono spesso selezionati in vitro attraverso tecniche di visualizzazione: dei fagi (phage display), dei ribosomi, del lievito oppure dell'mRNA . Le proteine leganti artificiali sono molto più piccole degli anticorpi (di solito meno di 100 residui amminoacidici), hanno una forte stabilità, mancano di legami disolfuro e possono essere espresse in alta resa in ambienti cellulari riducenti come il citoplasma batterico, al contrario degli anticorpi e dei loro derivati[18][19]. Sono quindi particolarmente adatti per produrre biosensori[20][21].
Interazioni enzimatiche
[modifica | modifica wikitesto]Le capacità di legame specifiche e l'attività catalitica degli enzimi li rendono biorecettori popolari. Il riconoscimento dell'analita è consentito attraverso diversi possibili meccanismi: 1) l'enzima converte l'analita in un prodotto rilevabile dal sensore, 2) rileva l'inibizione o l'attivazione dell'enzima da parte dell'analita o 3) monitora la modifica delle proprietà dell'enzima risultanti dall'interazione con l'analita .[15] Le ragioni principali per l'uso comune degli enzimi nei biosensori sono: 1) capacità di catalizzare un gran numero di reazioni; 2) potenziale per rilevare un gruppo di analiti (substrati, prodotti, inibitori e modulatori dell'attività catalitica); e 3) idoneità con diversi metodi di trasduzione per rilevare l'analita. In particolare, poiché gli enzimi non vengono consumati nelle reazioni, il biosensore può essere facilmente utilizzato in modo continuo. L'attività catalitica degli enzimi consente inoltre limiti di rilevamento inferiori rispetto alle comuni tecniche di legame. Tuttavia, la durata del sensore è limitata dalla stabilità dell'enzima.
Recettori di legame di affinita'
[modifica | modifica wikitesto]Gli anticorpi hanno un'elevata costante di legame superiore a 10^8 L/mol, che rappresenta un'associazione quasi irreversibile una volta che si è formata la coppia antigene-anticorpo. Per alcune molecole di analita come le proteine di legame di affinità del glucosio esistono che legano il loro ligando con un'elevata specificità come un anticorpo, ma con una costante di legame molto più piccola dell'ordine da 10^2 a 10^4 L/mol.
L'associazione tra analita e recettore è quindi di natura reversibile, e accanto ai due elementi accoppiati ci sono anche molecole di analita e recettore libere in una concentrazione misurabile. Nel caso del glucosio, ad esempio, la concanavalina A può funzionare come recettore di affinità esibendo una costante di legame di 4x10^2 L/mol.[22] L'uso di recettori di legame di affinità per scopi di biorilevamento è stato proposto da Schultz e Sims nel 1979[23] ed è stato successivamente configurato in un test fluorescente per misurare il glucosio nel relativo intervallo fisiologico compreso tra 4,4 e 6,1 mmol/l.[24] Il principio del sensore ha il vantaggio di non consumare l'analita in una reazione chimica come accade nei saggi enzimatici.
Interazioni degli acidi nucleici
[modifica | modifica wikitesto]I biosensori che usano recettori a base di acido nucleico possono essere basati su interazioni di accoppiamento di basi complementari denominate genosensori o su specifici riproduzioni di anticorpi a base di acido nucleico (aptameri) come aptasensori.[25] Nel primo, il processo di riconoscimento si basa sul principio dell'accoppiamento di basi complementari, adenina:timina e citosina:guanina nel DNA . Se la sequenza di acido nucleico bersaglio è nota, le sequenze complementari possono essere sintetizzate, etichettate e quindi immobilizzate sul sensore. L'evento di ibridazione può essere rilevato otticamente e la presenza di DNA/RNA bersaglio può essere accertata. In quest'ultimo, gli aptameri generati contro il bersaglio lo riconoscono attraverso l'interazione di specifiche interazioni non covalenti e tramite l'adattamento indotto. Questi aptameri possono essere etichettati facilmente con un fluoroforo/nanoparticelle metalliche per il rilevamento ottico o possono essere impiegati per piattaforme di rilevamento elettrochimiche o a rimozione senza etichetta per un'ampia gamma di molecole bersaglio o bersagli complessi come cellule e virus.[26][27] Inoltre, gli aptameri possono essere combinati con enzimi dell'acido nucleico, come DNAzimi che scindono l'RNA, fornendo sia il riconoscimento del bersaglio che la generazione del segnale in una singola molecola, che mostra potenziali applicazioni nello sviluppo di biosensori multiplex.[28]
Epigenetica
[modifica | modifica wikitesto]È stato proposto che i risonatori ottici integrati opportunamente ottimizzati possano essere sfruttati per rilevare modificazioni epigenetiche (es metilazione del DNA, modificazioni post-traduzionali dell'istone) nei fluidi corporei di pazienti affetti da cancro o altre malattie.[29] Al giorno d'oggi, a livello di ricerca, vengono sviluppati biosensori fotonici con ultrasensibilità per rilevare facilmente le cellule cancerose all'interno dell'urina del paziente.[30] Diversi progetti di ricerca mirano a sviluppare nuovi dispositivi portatili che utilizzino cartucce usa e getta economiche, ecocompatibili che richiedono solo una semplice manipolazione senza bisogno di ulteriori elaborazioni, lavaggi o manipolazioni da parte di tecnici esperti.[31]
Organelli
[modifica | modifica wikitesto]Gli organelli formano compartimenti separati all'interno delle cellule e di solito svolgono funzioni in modo indipendente. Diversi tipi di organelli hanno varie vie metaboliche e contengono enzimi per svolgere la loro funzione. Gli organelli comunemente usati includono lisosoma, cloroplasto e mitocondri.
Il modello di distribuzione spazio-temporale del calcio è strettamente correlato alla via di segnalazione onnipresente. I mitocondri partecipano attivamente al metabolismo degli ioni calcio per controllare la funzione e anche modulare le vie di segnalazione legate al calcio. Gli esperimenti hanno dimostrato che i mitocondri hanno la capacità di rispondere alle alte concentrazioni di calcio generate in loro prossimità aprendo i canali del calcio.[32] In questo modo, i mitocondri possono essere utilizzati per rilevare la concentrazione di calcio nel mezzo e il rilevamento è molto sensibile a causa dell'elevata risoluzione spaziale. Un'altra applicazione dei mitocondri viene utilizzata per il rilevamento dell'inquinamento dell'acqua. La tossicità dei composti detergenti danneggerà la cellula e la struttura subcellulare, compresi i mitocondri. I detersivi provocheranno un effetto rigonfiante che potrebbe essere misurato da una variazione dell'assorbanza. I dati dell'esperimento mostrano che il tasso di variazione è proporzionale alla concentrazione di detersivo, fornendo uno standard elevato per la precisione del rilevamento.[33]
Cellule
[modifica | modifica wikitesto]Le cellule sono spesso utilizzate nei biorecettori perché sono sensibili all'ambiente circostante e possono rispondere a tutti i tipi di stimolanti. Le cellule tendono ad attaccarsi alla superficie in modo da poter essere facilmente immobilizzate. Rispetto agli organelli rimangono attivi per un periodo più lungo e la riproducibilità li rende riutilizzabili. Sono comunemente usati per rilevare parametri globali come condizioni di stress, tossicità e derivati organici. Possono anche essere usati per monitorare l'effetto del trattamento dei farmaci. Un'applicazione consiste nell'utilizzare le cellule per determinare gli erbicidi che sono il principale contaminante acquatico.[34] Le microalghe vengono intrappolate su una microfibra di quarzo e la fluorescenza della clorofilla modificata dagli erbicidi viene raccolta sulla punta di un fascio di fibre ottiche e trasmessa ad un fluorimetro. Le alghe vengono continuamente coltivate per ottenere misurazioni ottimizzate. I risultati mostrano che il limite di rilevamento di alcuni erbicidi può raggiungere un livello di concentrazione inferiore a ppb. Alcune cellule possono essere utilizzate anche per monitorare la corrosione microbica.[35] Pseudomonas sp. viene isolato dalla superficie del materiale corroso e immobilizzato su una membrana di acetilcellulosa. L'attività respiratoria è determinata misurando il consumo di ossigeno. Esiste una relazione lineare tra la corrente generata e la concentrazione di acido solforico. Il tempo di risposta è correlato al caricamento delle celle e degli ambienti circostanti e può essere controllato a non più di 5 minuti.
Tessuto
[modifica | modifica wikitesto]I tessuti vengono utilizzati per il biosensore per l'abbondanza di enzimi esistenti. I vantaggi dei tessuti come biosensori includono quanto segue:[36]
- easier to immobilize compared to cells and organelles
- the higher activity and stability from maintaining enzymes in the natural environment
- the availability and low price
- the avoidance of tedious work of extraction, centrifuge, and purification of enzymes
- necessary cofactors for an enzyme to function exists
- the diversity providing a wide range of choices concerning different objectives.
There also exist some disadvantages of tissues, like the lack of specificity due to the interference of other enzymes and longer response time due to the transport barrier.
Microbial biosensors exploit the response of bacteria to a given substance. For example, arsenic can be detected using the ars operon found in several bacterial taxon.[37]
Attaccamento superficiale degli elementi biologici
[modifica | modifica wikitesto]
Una parte importante di un biosensore consiste nel fissare gli elementi biologici (piccole molecole/proteine/cellule) alla superficie del sensore (che si tratti di metallo, polimero o vetro). Il modo più semplice è funzionalizzare la superficie per rivestirla con gli elementi biologici. Questo può essere fatto da polilisina, aminosilano, epossisilano o nitrocellulosa nel caso di chip di silicio/vetro di silice. Successivamente, l'agente biologico legato può anche essere fissato, ad esempio mediante deposizione strato per strato di rivestimenti polimerici con carica alternata.[38]
In alternativa, è possibile utilizzare reticoli tridimensionali ( idrogel / xerogel ) per intrappolarli chimicamente o fisicamente (per cui chimicamente intrappolato si intende che l'elemento biologico è mantenuto in posizione da un forte legame, mentre fisicamente sono mantenuti in posizione non essendo in grado di passare attraverso i pori della matrice di gel). L'idrogel più comunemente usato è il sol-gel, silice vetrosa generata dalla polimerizzazione di monomeri di silicato (aggiunto come tetraalchil ortosilicati, come TMOS o TEOS) in presenza degli elementi biologici (insieme ad altri polimeri stabilizzanti, come il PEG) in il caso di intrappolamento fisico.[39]
Un altro gruppo di idrogel, che si fissano in condizioni adatte per cellule o proteine, sono l'idrogel di acrilato, che polimerizza dopo l'inizio dei radicali . Un tipo di iniziatore radicalico è un radicale perossido, tipicamente generato combinando un persolfato con TEMED (i gel di poliacrilammide sono anche comunemente usati per l'elettroforesi delle proteine ),[40] in alternativa la luce può essere usata in combinazione con un fotoiniziatore, come il DMPA ( 2, 2-dimetossi-2-fenilacetofenone ).[41] I materiali intelligenti che imitano i componenti biologici di un sensore possono anche essere classificati come biosensori utilizzando solo il sito attivo o catalitico o configurazioni analoghe di una biomolecola.[42]
Biotrasduttore
[modifica | modifica wikitesto]
I biosensori possono essere classificati in base al tipo di biotrasduttore . I tipi più comuni di biotrasduttori utilizzati nei biosensori sono:
- biosensori elettrochimici
- biosensori ottici
- biosensori elettronici
- biosensori piezoelettrici
- biosensori gravimetrici
- biosensori piroelettrici
- biosensori magnetici
Elettrochimico
[modifica | modifica wikitesto]I biosensori elettrochimici sono normalmente basati sulla catalisi enzimatica di una reazione che produce o consuma elettroni (tali enzimi sono giustamente chiamati enzimi redox). Il substrato del sensore di solito contiene tre elettrodi; un elettrodo di riferimento, un elettrodo di lavoro e un controelettrodo. L'analita target è coinvolto nella reazione che ha luogo sulla superficie dell'elettrodo attivo e la reazione può causare il trasferimento di elettroni attraverso il doppio strato (producendo una corrente) o può contribuire al potenziale del doppio strato (producendo una tensione). Possiamo misurare la corrente (la velocità del flusso di elettroni è ora proporzionale alla concentrazione dell'analita) a un potenziale fisso o il potenziale può essere misurato a corrente zero (questo fornisce una risposta logaritmica). Si noti che il potenziale dell'elettrodo di lavoro o attivo è sensibile alla carica spaziale e questo viene spesso utilizzato. Inoltre, il rilevamento elettrico diretto e senza etichetta di piccoli peptidi e proteine è possibile grazie alle loro cariche intrinseche utilizzando transistor ad effetto di campo sensibili agli ioni biofunzionalizzati .[43]
Un altro esempio, il biosensore potenziometrico, (potenziale prodotto a corrente zero) fornisce una risposta logaritmica con un'elevata gamma dinamica. Tali biosensori sono spesso realizzati serigrafando i modelli degli elettrodi su un substrato di plastica, rivestito con un polimero conduttore e quindi viene attaccata una proteina (enzima o anticorpo). Hanno solo due elettrodi e sono estremamente sensibili e robusti. Consentono il rilevamento di analiti a livelli precedentemente ottenibili solo da HPLC e LC/MS e senza una rigorosa preparazione del campione. Tutti i biosensori di solito comportano una preparazione minima del campione poiché il componente di rilevamento biologico è altamente selettivo per l'analita in questione. Il segnale è prodotto da cambiamenti elettrochimici e fisici nello strato di polimero conduttore dovuti a cambiamenti che si verificano sulla superficie del sensore. Tali cambiamenti possono essere attribuiti alla forza ionica, al pH, all'idratazione e alle reazioni redox, quest'ultime dovute al turn over dell'enzima che reagisce con il substrato.[44] I transistor ad effetto di campo, in cui la regione di gate è stata modificata con un enzima o un anticorpo, possono anche rilevare concentrazioni molto basse di vari analiti poiché il legame dell'analita alla regione di gate del FET provoca un cambiamento nella corrente di drain-source.
Lo sviluppo di biosensori basati sulla spettroscopia di impedenza sta guadagnando terreno al giorno d'oggi e molti di questi dispositivi/sviluppi si trovano nel mondo accademico e nell'industria. Uno di questi dispositivi, basato su una cella elettrochimica a 4 elettrodi, che utilizza una membrana di allumina nanoporosa, ha dimostrato di rilevare basse concentrazioni di alfa trombina umana in presenza di un elevato background di albumina sierica.[45] Anche gli elettrodi interdigitati sono stati utilizzati per i biosensori di impedenza.[46]
Interruttore di canale ionico
[modifica | modifica wikitesto]

È stato dimostrato che l'uso di canali ionici offre un rilevamento altamente sensibile delle molecole biologiche bersaglio.[47] Incorporando i canali ionici in membrane a doppio strato supportate o legate (t-BLM) attaccate a un elettrodo d'oro, viene creato un circuito elettrico. Le molecole di cattura come gli anticorpi possono essere legate al canale ionico in modo che il legame della molecola bersaglio controlli il flusso ionico attraverso il canale. Questo si traduce in un cambiamento misurabile nella conduzione elettrica che è proporzionale alla concentrazione dell'analita target.
Un biosensore di interruttore del canale ionico (ICS) può essere creato utilizzando la gramicidina, un canale peptidico dimerico, in una membrana a doppio strato legata.[48] Un peptide di gramicidina, con annesso anticorpo, è mobile e uno è fisso. La rottura del dimero interrompe la corrente ionica attraverso la membrana. L'entità della variazione del segnale elettrico è notevolmente aumentata separando la membrana dalla superficie metallica utilizzando un distanziatore idrofilo.
Il rilevamento quantitativo di un'ampia classe di specie bersaglio, tra cui proteine, batteri, farmaci e tossine è stato dimostrato utilizzando diverse configurazioni di membrana e di cattura.[49][50] Il progetto di ricerca europeo Greensense sviluppa un biosensore per eseguire lo screening quantitativo di sostanze d'abuso come THC, morfina e cocaina[51] nella saliva e nelle urine.
Note
[modifica | modifica wikitesto]- ^ a b vol. 249, 2017, DOI:10.1016/j.snb.2017.04.088, https://oadoi.org/10.1016/j.snb.2017.04.088.
- ^ 1987, p. 770, ISBN 978-0198547242.
- ^ 2012, p. 576, ISBN 9781118354230.
- ^ vol. 31, DOI:10.1002/adma.201806739, ISSN 0935-9648, PMID 31094032, https://oadoi.org/10.1002/adma.201806739.
- ^ vol. 8, 2008, DOI:10.3390/s8052932, PMID 27879858, https://oadoi.org/10.3390/s8052932.
- ^ vol. 1, 2019, DOI:10.1039/C9NA00153K, https://oadoi.org/10.1039/C9NA00153K.
- ^ A. Hierlemann, O. Brand, C. Hagleitner, H. Baltes, "Microfabrication techniques for chemical/biosensors", Proceedings of the IEEE 91 (6), 2003, 839–863.
- ^ A. Hierlemann, H. Baltes, "CMOS-based chemical microsensors", The Analyst 128 (1), 2003, pp. 15–28.
- ^ clemson.edu, https://web.archive.org/web/20170102201924/http://www.clemson.edu/c3b/biosensorsPrimer.html. URL consultato il 28 gennaio 2013 (archiviato dall'url originale il 2 gennaio 2017).
- ^ vol. 35, DOI:10.1016/j.tibtech.2017.03.013, PMID 28456344, https://oadoi.org/10.1016/j.tibtech.2017.03.013.
- ^ Highly sensitive electrochemiluminescence detection of a prostate cancer biomarker, in J. Mater. Chem. B, vol. 5, n. 32, 2017, pp. 6681–6687, DOI:10.1039/c7tb01557g, PMID 32264431.
- ^ Biosensors and biochips: Advances in biological and medical diagnostics, in Fresenius' Journal of Analytical Chemistry, vol. 366, 6–7, 2000, pp. 540–551, DOI:10.1007/s002160051549, PMID 11225766.
- ^ An electrochemiluminescencesupramolecular approach to sarcosine detection for early diagnosis of prostate cancer, in Faraday Discuss, vol. 185, 2015, pp. 299–309, DOI:10.1039/c5fd00096c, PMID 26394608.
- ^ High-performance graphene-based biosensor using a metasurface of asymmetric silicon disks, in IEEE Sensors Journal, vol. 22, n. 3, 2022, pp. 2037–2044, DOI:10.1109/JSEN.2021.3134205.
- ^ a b Fiber-optic biosensors – an overview, in Analytical and Bioanalytical Chemistry, vol. 372, 5–6, 2002, pp. 664–682, DOI:10.1007/s00216-002-1235-9, PMID 11941437.
- ^ Role of Serology in the Coronavirus Disease 2019 Pandemic, in Clinical Infectious Diseases, vol. 71, n. 8, pp. 1935–1936, DOI:10.1093/cid/ciaa510, PMID 32357206.
- ^ Aptamers, antibody scFv, and antibody Fab' fragments: An overview and comparison of three of the most versatile biosensor biorecognition elements, in Biosens Bioelectron, vol. 85, Nov 2016, pp. 32–45, DOI:10.1016/j.bios.2016.04.091, PMID 27155114.
- ^ Non-immunoglobulin scaffolds: a focus on their targets, in Trends Biotechnol, vol. 33, Jul 2015, pp. 408–418, DOI:10.1016/j.tibtech.2015.03.012, PMID 25931178.
- ^ Engineered proteins with desired specificity: DARPins, other alternative scaffolds and bispecific IgGs, in Curr Opin Struct Biol, vol. 27, Aug 2014, pp. 102–112, DOI:10.1016/j.sbi.2014.05.011, PMID 25033247.
- ^ Knowledge-based design of reagentless fluorescent biosensors from a designed ankyrin repeat protein, in Protein Eng Des Sel, vol. 23, Apr 2010, pp. 229–241, DOI:10.1093/protein/gzp074, PMID 19945965.
- ^ Reagentless fluorescent biosensors from artificial families of antigen binding proteins, in Biosens Bioelectron, vol. 26, Jun 2011, pp. 4184–4190, DOI:10.1016/j.bios.2011.04.030, PMID 21565483.
- ^ J. S. Schultz, Affinity sensor: A New Technique for Developing Implantable Sensors for Glucose and Other Metabolites, in Diabetes Care, vol. 5, n. 3, 1982, pp. 245–253, DOI:10.2337/diacare.5.3.245, PMID 6184210.
- ^ J. S. Schultz, Affinity sensors for individual metabolites, in Biotechnol. Bioeng. Symp., vol. 9, n. 9, 1979, pp. 65–71, PMID 94999.
- ^ R. Ballerstadt, A Fluorescence Affinity Hollow Fiber Sensor for Continuous Transdermal Glucose Monitoring, in Anal. Chem., vol. 72, n. 17, 2000, pp. 4185–4192, DOI:10.1021/ac000215r, PMID 10994982.
- ^ Nanomaterial based aptasensors for clinical and environmental diagnostic applications, in Nanoscale Advances, vol. 1, n. 6, pp. 2123–2138, DOI:10.1039/C9NA00153K.
- ^ Development of DNA aptamers using Cell-SELEX, in Nature Protocols, vol. 5, n. 6, 2010, pp. 1169–1185, DOI:10.1038/nprot.2010.66, PMID 20539292.
- ^ Microtitre Plate Based Cell-SELEX Method, in Bio-Protocol, vol. 8, n. 20, pp. e3051, DOI:10.21769/BioProtoc.3051, PMID 34532522.
- ^ DNA-only bioassay for simultaneous detection of proteins and nucleic acids, in Analytical and Bioanalytical Chemistry, vol. 413, n. 20, 2021, pp. 4925–4937, DOI:10.1007/s00216-021-03458-6, PMID 34184101.
- ^ vol. 4, DOI:10.1002/jbio.201000123, PMID 21567973, https://oadoi.org/10.1002/jbio.201000123.
- ^ vol. 1, DOI:10.1515/nanoph-2012-0021, PMID 26918228, https://oadoi.org/10.1515/nanoph-2012-0021.
- ^ GLAM Project - Glass-Laser Multiplexed Biosensor, https://web.archive.org/web/20160214015129/http://www.glam-project.eu/. URL consultato il 30 ottobre 2022 (archiviato dall'url originale il 14 febbraio 2016).
- ^ Mitochondria as biosensors of calcium microdomains, in Cell Calcium, vol. 26, n. 5, 1999, pp. 193–199, DOI:10.1054/ceca.1999.0076, PMID 10643557.
- ^ The Mitochondria as Biosensors for the Monitoring of Detergent Compounds in Solution, in Analytical Biochemistry, vol. 292, n. 2, 2001, pp. 305–307, DOI:10.1006/abio.2001.5097, PMID 11355867.
- ^ Optical whole-cell biosensor using Chlorella vulgaris designed for monitoring herbicides, in Biosensors & Bioelectronics, vol. 18, n. 4, 2003, pp. 457–63, DOI:10.1016/s0956-5663(02)00157-4, PMID 12604263.
- ^ Microbial corrosion monitoring by an amperometric microbial biosensor developed using whole cell of Pseudomonas sp., in Biosensors & Bioelectronics, vol. 16, 9–12, 2001, pp. 995–1000, DOI:10.1016/s0956-5663(01)00203-2, PMID 11679280.
- ^ Plant tissue-and photosynthesis-based biosensors, in Biotechnology Advances, vol. 26, n. 4, 2008, pp. 370–378, DOI:10.1016/j.biotechadv.2008.04.001, PMID 18495408.
- ^ Construction and use of broad host range mercury and arsenite sensor plasmids in the soil bacterium Pseudomonas fluorescens OS8, in Microbial Ecology, vol. 41, n. 4, 2001, pp. 360–368, DOI:10.1007/s002480000095, PMID 12032610.
- ^ Nanomedicine and its potential in diabetes research and practice, in Diabetes Metab Res Rev, vol. 24, n. 8, 2008, pp. 604–10, DOI:10.1002/dmrr.893, PMID 18802934.
- ^ vol. 22, DOI:10.1016/j.bios.2006.12.025, PMID 17291744, https://oadoi.org/10.1016/j.bios.2006.12.025.
- ^ vol. 71, DOI:10.1021/ac990630n, PMID 10565275, https://oadoi.org/10.1021/ac990630n.
- ^ vol. 23, DOI:10.1016/j.bios.2008.01.012, PMID 18304798, https://oadoi.org/10.1016/j.bios.2008.01.012.
- ^ technologyreview.com, http://www.technologyreview.com/biomedicine/21603/?a=f.
- ^ Field Effect of Screened Charges: Electrical Detection of Peptides and Proteins by a Thin Film Resistor, in ChemPhysChem, vol. 7, n. 2, 2006, pp. 379–384, DOI:10.1002/cphc.200500484, PMID 16404758.
- ^ Copia archiviata, su universalsensors.co.uk. URL consultato il 30 ottobre 2022 (archiviato dall'url originale il 18 dicembre 2014).
- ^ vol. 126, 2019, DOI:10.1016/j.bios.2018.10.010, PMID 30396022, https://oadoi.org/10.1016/j.bios.2018.10.010.
- ^ vol. 204, 2014, DOI:10.1016/j.snb.2014.06.141, https://oadoi.org/10.1016/j.snb.2014.06.141.
- ^ 2005, pp. 608–610, DOI:10.1109/icsens.2005.1597772, ISBN 978-0-7803-9056-0.
- ^ Cornell BA, A biosensor that uses ion-channel switches, in Nature, vol. 387, n. 6633, 1997, pp. 580–583, DOI:10.1038/42432, PMID 9177344.
- ^ Oh S, Rapid detection of influenza A virus in clinical samples using an ion channel switch biosensor, in Biosensors & Bioelectronics, vol. 23, n. 7, 2008, pp. 1161–1165, DOI:10.1016/j.bios.2007.10.011, PMID 18054481.
- ^ Ion Channel Biosensors Part I Construction Operation and Clinical Studies, in IEEE Transactions on Nanotechnology, vol. 9, n. 3, 2010, pp. 313–322, DOI:10.1109/TNANO.2010.2041466.
- ^ Copia archiviata, su greensense-project.eu. URL consultato il 30 ottobre 2022 (archiviato dall'url originale il 16 maggio 2022).
Bibliografia
[modifica | modifica wikitesto]- Frieder Scheller, 1989, ISBN 978-3-05-500659-3.
- Massimo Grattarola, 1998, ISBN 978-0070031746.
- Scratching at the surface of biosensors – an Instant Insight discussing how surface chemistry lets porous silicon biosensors fulfil their promise from the Royal Society of Chemistry
| Controllo di autorità | Thesaurus BNCF 36727 · LCCN (EN) sh85014260 · GND (DE) 4193016-2 · BNF (FR) cb12125424b (data) · J9U (EN, HE) 987007282392505171 |
|---|